RECCAM - Seagrass Meadows resilience to global warming: an analysis based on responses at ecophysiological, population and ecosystem levels
Sommario
Il cambiamento climatico dovrebbe causare alterazioni significative nel contesto globale, con effetti chiari e specifici negli oceani. Il Mar Mediterraneo è un ottimo modello per lo studio di tali effetti sugli ecosistemi marini. Praterie di seagrasses, ed in particolare quelli dominati da Posidonia oceanica e Cymodocea nodosa, sono tra gli habitat più minacciati del Mediterraneo. Questo progetto ha lo scopo di contribuire alla comprensione dei principali meccanismi di risposta di questi habitat chiave al riscaldamento globale, probabilmente la componente principale del cambiamento climatico. A tal fine, vengono perseguiti tre approcci, alcuni relativamente inesplorati ma cruciali per ottenere una conoscenza adeguata degli impatti di temperatura in aumento. In primo luogo, studieremo la tolleranza fisiologica delle specie di fanerogame marine mediterranee a stress termico. In secondo luogo, valuteremo l'effetto di riscaldamento sull’erbivorismo. In terzo luogo, analizzeremo le interazioni tra cambiamento climatico e altri fattori di stress, in particolare l'eutrofizzazione ed i disturbi meccanici.
Ruolo SZN
Istituzione partecipante per la caratterizzazione genetica di piante in situ e nei mesocosmi e per la valutazione dell'espressione genica in situ ed in condizioni controllate.
Partners
University of Barcelona, Spain; Spanish Oceanographic Institute (IEO), Spain; Stazione Zoologica Anton Dohrn, Italy; CSIC-CEAB, Spain
Durata
2014-2016
P. I.
Gabriele Procaccini
Project coordinator: Javier Romero Martinengo (University of Barcelona, Spain)
Istituzione finanziatrice
Ministerio de Economía y Competitividad (MINECO) - Spain
Contributo alla SZN
€ 16.000
Finanziato dal Programma Operativo ENPI CBC Mediterranean Sea Basin 2007/2013 dell’Unione Europea
Responsabile Scientifico Raffaella Casotti
Il progetto è indirizzato a fornire il necessario sostegno tecnico e organizzativo, soprattutto alle piccole e medie imprese (esistenti e potenziali) che operano nel settore agro-alimentare e in altri settori tradizionali del turismo, per la promozione di azioni coordinate volte ad aumentare la quota dei proventi del turismo che avvantaggia le economie dei paesi del Mediterraneo. Il risultato principale del progetto riguarda la creazione di nuove attività imprenditoriali legate al turismo sostenibile in quattro aree costiere dotate di notevoli risorse naturali e culturali e il potenziamento di alleanze di imprese attraverso l'adozione di un approccio di gestione pubblica/privata, standard di qualità e incentivare sistemi in cui sono accoppiati gli obiettivi di sviluppo economico con la protezione e la valorizzazione del patrimonio naturale/culturale.
Inoltre, il progetto creerà una rete internazionale di destinazioni di turismo costiero sostenibile nel bacino del Mar Mediterraneo come luogo per lo sviluppo di metodologie comuni durante l’attuazione del progetto e nel lungo periodo quali il monitoraggio e la piattaforma promozionale partecipata da utilizzatori locali, nazionali e scientifici e aperto ai nuovi soggetti, che potrebbe stimolare ulteriori sviluppi di turismo costiero sostenibile sulla rotta del Mediterraneo.
Siamo partner del progetto ed il nostro ruolo è di creare il sistema di monitoraggio dei tre siti al fine di a) fornire dati in tempo reale sulle condizioni ambientali b) sensibilizzare i visitatori alle tematiche ambientali, diffondendo la cultura sceintifica anche attraverso azioni di "citizen science" c) formare gli operatori turistici affinchè siano coscienti del valore ambientale dei loro siti e partecipino alla loro valorizzazione e conservazione
Partners: il Ministero dei beni e delle attività culturali e del turismo (Coordinatore), la Penisola di Sinis (Sardegna), la Stazione Zoologica Anton Dohrm, la municipalità di Mahdia (Tunisia) e l’Istituto Nazionale di Scienze Marine e Tecnologia della Tunisia, la Al-Balqa Applied University e l’Area Marina protetta di Aqaba (Giordania)
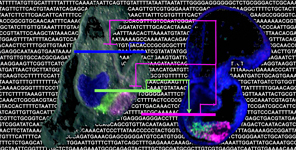
Gut patterning and PANcreas development in evolution and disease: a TRAnsCriptomic approach
Summary
Many genes that have been shown to cause diseases were originally identified because of their role in embryonic development, but were subsequently shown to be also important in the postnatal control of cell growth and differentiation. This is the case of many transcription factors (TF) among which the ParaHox gene Xlox, whose mammalian homolog, Pdx1, is well known for its role in specification of the pancreas, and subsequent formation and maintenance of pancreatic beta-cells. Pdx1 is a causal factor in the development of diabetes, wherein there is a deficiency in insulin production of beta-cells within the pancreas. Moreover, mis-expression of Pdx1 is commonly seen in intestinal disorders such as Crohn’s disease. Here we propose to combine analyses in the highly simple but phylogenetically relevant sea urchin embryo and sea star embryo models with developmentally targeted mouse transcriptome data to characterize regulatory connections that are downstream of the disease-related Xlox/Pdx1 transcription factor.
What we do
We are coordinator of the project and Operating unit SZN and will perform all manipulations and analyses in sea urchin and sea star embryos and all bioinformatic analyses and evolutionary comparisons.
Partners
Stazione Zoologica Anton Dohrn, Napoli; Laboratorio di Medicina Molecolare e Genomica, Università degli Studi di Salerno.
Research Area
Organismal Biology
Project Lifetime
April 2014 to December 2015
SZN Role
Coordinator
Principal Investigator
Funding Institution
MIUR Progetti Premiali (DLGS 213/99)
Contribution to SZN
€169.143,00 (MIUR contribution)
Publications
Annunziata R and Arnone MI (2014). A dynamic network of regulatory interactions explains ParaHox gene control of gut patterning in the sea urchin embryo. Development 141: 2462-72.
Perillo M, Wang YJ, Leach SD and Arnone MI. Specification and differentiation of pancreatic, acinar-like cells in the sea urchin embryo and larva. Submitted to Development.
Meet the team
Maria I. Arnone, primo ricercatore
Rossella Annunziata, postdoc
Claudia Cuomo, PhD student
Elijah Lowe, postdoc

Multidisciplinarity training in evo-devo and neurobiology of marine animal models
Summary
Neptune is a multidisciplinary training network in evo-devo and neurobiology of marine animal models. Through the use of advanced methods of genetic analysis and imaging technologies, Neptune aims at solving an array of important questions in the evolution, development, neurobiology and ecology of marine invertebrates.
Neptune is training a new generation of young researchers by combining the strengths of modern technologies with a real understanding of traditional approaches. The Neptune consortium involves seven academic institutions and one industrial partner that will provide Neptune fellows with expertise, specialized equipment and training on a wide range of approaches and methodologies incorporated in evolutionary developmental biology and marine neurobiology.
What we do
We are one of the seven partners and are contributing to the WP "Evolution of sensory systems" by studying photoreceptor evolution in echinoderms and hemichordates (Ambulacraria).
Partners
European Molecular Biology Laboratory, Heidelberg - DE; Stazione Zoologica Anton Dohrn, Napoli – IT; Uppsala University, Uppsala – SE; Max Plank Institute for Developmental Biology, Tübigen – DE; University College London, London – UK; Sars International Center for Marine Molecular Biology, Bergen – NO; Centre National de la Recherche Scientifique, Villefranches sur mer, Lion - FR; Associate industrial partner: ZEISS.
Research Area
Organismal Biology
Project Lifetime
March 2013 to February 2017
SZN Role
Partner
Principal Investigator
Funding Institution
European Commission, FP7 Call for Proposal: FP7-PEOPLE-2012-ITN
Marie Curie Action - Initial Training Network (ITN)
Grant no. 317172
Contribution to SZN
€302.697,45 (EU contribution)
Dedicated website
Media - Pictures

Publications
Valero-Gracia A, Petrone L, Oliveri P, Nilsson DE, Arnone MI. Non-directional Photoreceptors in the Pluteus of Strongylocentrotus purpuratus Frontiers in Ecology and Evolution (2016) 4, 127.
D’Aniello S, Delroisse J, Valero-Gracia A, Lowe EK, Byrne M, Cannon JT, Halanych KM, Elphick MR, Mallefet J, Kaul-Strehlow S, Lowe CJ, Flammang P, Ullrich-Lüter E, Wanninger A and Arnone MI (2015). Opsin evolution in the Ambulacraria. Marine Genomics, 24: 177-183.
Ullrich-Lüter E, D’Aniello S and Arnone MI (2013). C-opsin expressing photoreceptors in echinoderms. Integr Comp Biol 53: 27-38.
Peterson KJ, Su Y-H, Arnone MI, Swalla B, and King B (2013). microRNAs Support the Monophyly of Enteropneust Hemichordates. J Exp Zool B Mol Dev Evol 320: 368-374.
Meet the team
Maria I. Arnone, primo ricercatore
Alberto Valero Gracia, PhD student
Marie Curie Career Integration Grant (FP7-PEOPLE-2011-CIG)
Summary
The main interest of the project is to study the Nitric Oxide Synthase (NOS) family evolution and its regulation during amphioxus development. In the framework of the project, setting-up an amphioxus facility at the SZN was a priority of national interest, representing the first Italian laboratory working with live amphioxus embryos on demand.
Since Furchgott, Ignarro and Murad won the Nobel Prize in Medicine or Physiology in 1998 for their breakthrough work on the role of nitric oxide (NO) as a multifunctional signaling molecule, many reports have shown the seemingly limitless range of body functions controlled by this compound. To manipulate the endogenous NO level for therapeutic benefits using NOS gene therapy is essential to understand the physiological and developmental functions of different NOS isoforms (nNOS, iNOS, eNOS). Due to their extensive conservation over evolutionary time, one would expect greater differences and structural changes in NOS genes than that we have observed (Andreakis 2011), reflecting the very ancient and essential nature of Nitric Oxide biological pathways.
Surprisingly, a single molecule, identical in all living animals, can fulfil a huge range of different functions. This suggests that differences in the regulation of NOS enzymes expression are key in explaining their functional diversification, functional novelties and degree of complexity.
What we do
We use as animal model system the cephalochordate amphioxus Branchiostoma lanceolatum, from the Gulf of Napoli (Italy) and from Banyuls-sur-Mer (France), with comparative and multidisciplinary approaches in the field of Evolutionary and Developmental Biology (Evo-Devo). The primary aim of this project is to perform a detailed study of the duplicated set of NOS genes during amphioxus development, trying to establish the basic primary NOS roles that are evolutionary conserved in chordates.
Partners
Stazione Zoologica Anton Dohrn, Napoli.
Research Area
Organismal Biology
Project Lifetime
August 2011 to July 2015
SZN Role
Coordinator
Principal Investigator
Funding Institution
European Commission, FP7 Call for Proposal: FP7-PEOPLE-2011-CIG
Grant no. 293871
Contribution to SZN
€100.000 (EU contribution)
Dedicated website
http://cordis.europa.eu/project/rcn/99685_en.html
Publications
Coppola U, Annona G, D’Aniello S* and Ristoratore F* (2015). Rab32/38 duplicated genes in chordate pigmentation: an evolutionary perspective BMC Evolutionary Biology, under review.
Annona G, Holland ND* and D’Aniello S* (2015). Evolution of the notochord. EvoDevo, in press.
Anishchenko E and D’Aniello S* (2015). Tunicate neurogenesis: the case of the SoxB2 missing CNE. Mathematical Models in Biology (Springer), in press.
Vassalli QA, Anishchenko E, Caputi L, Sordino P, D'Aniello S* and Locascio A* (2015). Regulatory elements retained during chordate evolution: Coming across tunicates. Genesis 53: 66-81.
Pascual-Anaya J, D’Aniello S, Kuratani S and Garcia-Fernàndez J (2013). Evolution of the Hox clusters in deuterostomes. BMC Developmental Biology 13: 26.
Meet the team
Salvatore D’Aniello, Ricercatore
Evgeniya Anishchenko, post-doc
Giovanni Annona, post-doc
Filomena Caccavale, PhD student