L’area di Elettroforesi e PCR è attrezzata con 9 macchine per PCR e con la strumentazione necessaria alla preparazione di gel di agarosio, alla corsa elettroforetica, all’acquisizione di immagini UV, alla quantificazione di acidi nucleici (NanoDrop).
L’area di Elettroforesi e PCR è attrezzata con 9 macchine per PCR e con la strumentazione necessaria alla preparazione di gel di agarosio, alla corsa elettroforetica, all’acquisizione di immagini UV, alla quantificazione di acidi nucleici (NanoDrop).
 Responsabile: Alessandro Amoroso
Responsabile: Alessandro Amoroso
Questo indirizzo email è protetto dagli spambots. È necessario abilitare JavaScript per vederlo.
Interno #345
| Strumentazione prenotabile |  |
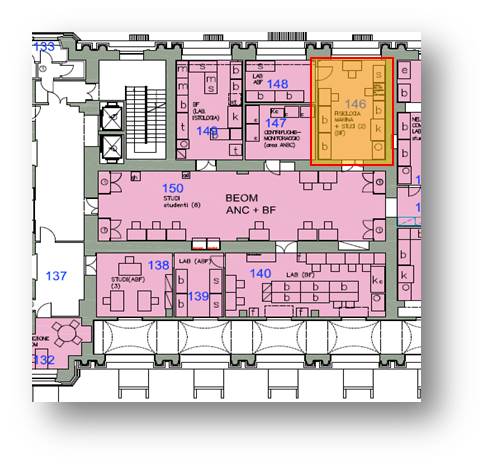
| Stanza #146 | |
| Cappa Chimica per la purificazione di acidi nucleici | |
 |
|
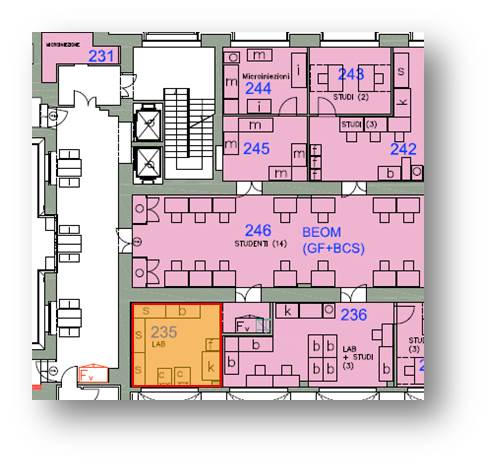
| Stanza #235 | |
| PCR HYBAID Express #1 | |
| PCR HYBAID Express #2 | |
| PCR HYBAID PX2 | |
| PCR BIORAD C1000 #1 | |
| PCR BIORAD C1000 #2 | |
| PCR MJResearch PTC 100 | |
| PCR MJResearch MiniCycler | |
| PCR GeneAmp System 9700 | |
| PCR EPPENDORF Mastercycler | |
| Bagnetto Termostatato JULABO SW22 | |
| Bagnetto Termostatato GFL | |
| Stufa per Ibridazione FINEPCR | |
| Stufa per Ibridazione TECNE |
|
Primo piano Ala EST, #146 |
Secondo piano Ala EST, #235 |
 |
 |
 L’area dedicata alla Centrifugazione e Ultracentrifugazione è dotata di diverse attrezzature che permettono la centrifugazione a varie velocità (fino a 90000 rpm nel caso dell’Ultracentrifugazione), con rotori ad angolo fisso o basculante, a temperatura controllata.
L’area dedicata alla Centrifugazione e Ultracentrifugazione è dotata di diverse attrezzature che permettono la centrifugazione a varie velocità (fino a 90000 rpm nel caso dell’Ultracentrifugazione), con rotori ad angolo fisso o basculante, a temperatura controllata.
 Responsabile: Alessandro Amoroso
Responsabile: Alessandro Amoroso
Questo indirizzo email è protetto dagli spambots. È necessario abilitare JavaScript per vederlo.
Interno #345
| Strumentazione prenotabile |
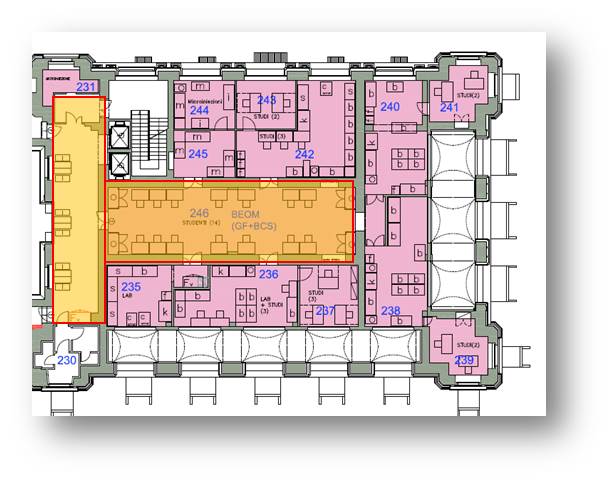
| Stanza #246 |
| Concentratore EPPENDORF 5301 |
| Centrifuga refrigerata 15/50mL EPPENDORF 5810R |
| Centrifuga refrigerata EPPENDORF 5430R |
| Incubatore Shaker INNOVA 4 |
| Transetto |
| Ultracentrifuga BECKMAN Optima XL 100K |
| Centrifuga BECKMAN AvantiJ25 |
| Centrifuga BECKMAN J2 MC |


|
Secondo Piano Stanza #246 e Transetto |
 |
 L’area di Estrazione e manipolazione di acidi nucleici una al primo piano e due al secondo piano dell’ala est. Questi laboratori sono attrezzati con cappe dedicate a diverse attività.
L’area di Estrazione e manipolazione di acidi nucleici una al primo piano e due al secondo piano dell’ala est. Questi laboratori sono attrezzati con cappe dedicate a diverse attività.
 Responsabile: Alessandro Amoroso
Responsabile: Alessandro Amoroso
Questo indirizzo email è protetto dagli spambots. È necessario abilitare JavaScript per vederlo.
Interno #345
| Strumentazione prenotabile | |
| Stanza #146 | |
| Cappa Chimica per la purificazione di acidi nucleici |  |
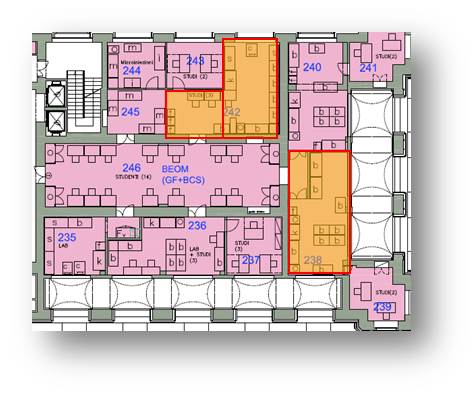
| Stanza #242 | |
|
Cappa Chimica dotata di pHmetro THERMO SCIENTIFIC Orion Star A211 per la preparazione di soluzioni |
 |
| Stanza #238 | |
| Cappa Chimica per la purificazione di acidi nucleici |  |
|
Primo piano Ala EST, #146 |
Secondo Piano Ala EST: Stanze # 242 e 238 |
 |
 |